Die Regulation der Transkription ist eine Schnittstelle zwischen Zellwachstum und HIV-stimulierter Genexpression
Forschungsbericht (importiert) 2007 - Max-Planck-Institut für molekulare Physiologie
Regulation der eukaryotischen Transkriptionselongation
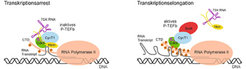
Die Transkription eukaryotischer Gene zur Produktion reifer mRNA-Transkripte ist ein komplexer Prozess, an dem neben der chromosomalen DNA eine Vielzahl von Proteinfaktoren beteiligt ist. Kurz nach der Transkriptionsinitiation kommt es zu einem Halt, der durch negative Faktoren vermittelt wird und als Kontrollpunkt vor dem Eintritt in die produktive Elongation dient. Die Wiederaufnahme der Transkription wird erst durch die Wirkung eines positiven Transkriptionselongationsfaktors (P-TEFb) möglich, der zu einer spezifischen Phosphorylierung der C-terminalen Domäne (CTD) der RNA-Polymerase II führt [1]. P-TEFb besteht aus der Cyclin-abhängigen Kinase Cdk9 und einem Cyclin T. Durch die Kinase werden die Serinreste an Position 2 der Heptadsequenzen hyper-phosphoryliert, was zur Dissoziation der negativen Regulationsfaktoren von der RNA-Polymerase II führt. Dadurch wird der Übergang in die produktive Transkriptionselongation eingeleitet zur Bildung vollständiger mRNA-Transkripte.
Die funktionale Bedeutung von P-TEFb für die eukaryotische Genexpression wurde zunächst im Jahre 1998 an der HIV-stimulierten Transkription erkannt. HIV-1 rekrutiert P-TEFb an die RNA-Polymerase II über die Assoziation mit dem viralen Transaktivatorprotein Tat und dem Promotor TAR-RNA-Element [2]. Das Virusprotein erzielt damit eine erhöhte Expression der viralen Gene gegenüber der basalen Transkriptionsaktivität. Erst fünf Jahre später wurde entdeckt, dass auch P-TEFb selbst reguliert wird und zusammen mit dem Protein Hexim1 und der kleinen nukleären RNA 7SK in einem inaktiven Komplex vorliegt [1]. Das 43 kiloDalton große Protein Hexim1 inhibiert die Kinaseaktivität von Cdk9 und ist so an der Regulation der CTD-Phosphorylierung beteiligt (Abb. 1). Einzelne spezifische Aktivatoren und zumindest ein genereller Chromatin-verändernder Faktor, das Bromodomäne 4-Protein (Brd4), rekrutieren dabei P-TEFb zu den Transkriptionskomplexen. Die Arbeitsgruppe von Matthias Geyer am MPI für molekulare Physiologie in Dortmund befasst sich mit den molekularen Mechanismen der Regulation der Transkriptionselongation und der Strukturanalyse des inhibierten sowie des transkriptionsaktiven P-TEFb-Komplexes.
Fehlregulationen von P-TEFb sind mit unterschiedlichen Krankheitsbildern verbunden
Die physiologische Bedeutung der Regulation von P-TEFb wurde in den vergangenen Jahren deutlich, als verschiedene Krankheitsbilder mit Fehlregulationen der Kinaseaktivität von Cdk9 assoziiert werden konnten. So spielt die Freisetzung und Aktivierung von P-TEFb in der Pathogenese der Herzmuskelhypertrophie eine wichtige Rolle. Diese Krankheit ist durch eine Vergrößerung der Herzmuskelzellen charakterisiert, die mit dem globalen Anstieg des RNA- und Proteingehalts in der Zelle verbunden ist. Ein unkontrollierter Zellwachstumsprozess wurde auch in vaskulären weichen Muskelzellen beobachtet, die mit Hexamethylenbisacetamid induziert wurden, was für das Hexim-Protein namensgebend war. Aufgrund seiner inhibitorischen Funktion wurde Hexim1 dabei als neues therapeutisches Ziel für die Behandlung von Arteriosklerose und Restinose identifiziert.
Parallel dazu wurde Hexim1 in einem onkologischen Zusammenhang als Inhibitor des Wachstums von Brustzellen identifiziert, der durch Östrogen herunterreguliert wird und in Brusttumoren abgeschwächt ist. Das Expressionslevel dieses zunächst als „estrogen down-regulated gene 1“ bezeichneten Proteins war in normalem Brustepithelzellgewebe signifikant höher gegenüber Tumorgeweben aus Brustzellen.
Schließlich stellt P-TEFb den Zielkomplex für das HIV-1-Transaktivator-Protein Tat dar. Tat bindet an die TAR-RNA-Promotorsequenz der langen terminalen Repeats der viralen Gene. Zusammen binden beide Moleküle die Cyclin-T1-Untereinheit des P-TEFb-Komplexes und aktivieren so die Transkriptionselongation mit einem bis heute unbekannten Mechanismus. Die viralen Gene werden dadurch um das bis zu 100fache häufiger transkribiert gegenüber der basalen Transkriptionsrate zellulärer Gene, was eine Voraussetzung für die Proteinproduktion zur Bildung neuer Viren darstellt.
Identifizierung der Cyclin T-bindenden Domäne in Hexim1
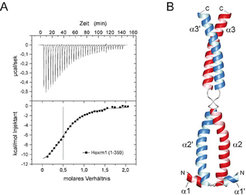
Biochemische Analysen der Interaktion von Cyclin T1 und Hexim1 führten zur Identifizierung der Cyclin T-bindenden Domäne (TBD) in Hexim1 als stabiles, C-terminales Fragment von 12,5 kDa Größe [3]. Die Strukturaufklärung mittels NMR-Spektroskopie zeigte dabei eine dimere, parallele Coiled-coil-Faltung aus zwei Segmenten, denen eine kurze Helix vorgelagert ist (Abb. 2), [4]. Messungen der Transkriptionsaktivität mit C-terminal verkürzten Hexim-Fragmenten in vivo sowie NMR-Kartierungsexperimente in vitro ergaben eine besondere Notwendigkeit des ersten Coiled-coil-Segments für die Interaktion mit Cyclin T1. In Mutagenesestudien konnte diese Region bestätigt und eine elektrostatisch getriebene Wechselwirkung zu Hexim1 ausgemacht werden. Die Stöchiometrie der Bindung zeigt darüber hinaus, dass ein Hexim1-Dimer mit nur einem Cyclin-T1-Molekül interagiert, wobei die Dissoziationskonstante der Bindungspartner im niedrigen mikromolaren Bereich liegt. Wie auf molekularer Ebene die Kinaseaktivität von Cdk9 durch Hexim1 inhibiert wird, das direkt nachweisbar nur Cyclin T1 bindet und so möglicherweise allosterisch wirkt, ist dabei noch ungeklärt. Hexim1 stellt damit neben den Kip- und Ink-Familien eine neue Klasse von proteinbasierten Inhibitoren der Cdk/Cyclin-Paare dar.
Die Cyclinbox-Domäne von Cyclin T1 besitzt eine regulatorische Helix
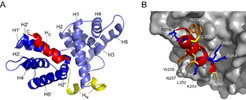
Die Struktur der Cyclinbox-Domäne von Cyclin T1 konnte im Komplex mit dem viralen Tat-Protein durch Röntgenkristallographie gelöst werden [5]. Dabei zeigt der Kernbereich dieser Domäne die erwartete Cyclinbox-Faltung, die sich aus zwei helikalen Wiederholungssequenzen (H1-H5 und H1'-H5') zusammensetzt (Abb. 3). Die Konformation der angrenzenden N- und C-terminalen Helices (HN und HC) zeigt jedoch Besonderheiten, die spezifisch für Cyclin T1 sind. Das Segment der Aminosäuren 250 bis 261 wurde schon in früheren Arbeiten als Tat/TAR-Erkennungsmotiv (TRM) beschrieben, das für die spezifische Bindung des HIV-1-Tat-Proteins an Cyclin T1 notwendig ist [2]. Dabei wurde angenommen, dass es zur Ausbildung eines intermolekularen Zinkfingers zwischen Tat und Cyclin T1 kommt, bei dem drei Aminosäuren aus Tat mit einem Cysteinrest an Position 261 in Cyclin T1 interagieren. Diese Vermutung wurde durch die Beobachtung bestärkt, dass das murine Cyclin T1 an gleicher Stelle kein Cystein besitzt, was unter anderem erklärt, warum es keine virale Variante von HIV bzw. SIV in Mäusen gibt.
Obwohl die Struktur des Tat-Proteins in dem Komplexkristall nicht hinreichend aufgelöst ist, zeigt sich, dass die TRM-Region der Cyclinbox in Cyclin T1 eine C-terminale Helix einnimmt, die in einer Spalte zwischen den beiden Wiederholungssequenzen liegt. Neben zwei positiven Ladungsresten ist insbesondere Cystein 261 an der Oberfläche des Moleküls zugänglich für Interaktionen. Weitere Reste, die möglicherweise die Bindung an das TAR-RNA-Promotorelement verstärken, würden erst durch einen Konformationswechsel dieser Helix im transkriptionsaktiven Komplex zugänglich.
Das HIV-1-Tat-Protein verdrängt Hexim1 zur Stimulation der viralen Transkription
Zur Generierung neuer Viruskopien in einer infizierten Zelle ist eine erhöhte Produktion der viralen Proteine notwendig. Das HI-Virus erreicht dies über das Transaktivatorprotein Tat, das die TAR-Promotor-RNA am 5'-Ende der viralen Transkripte an P-TEFb heranführt [2]. Untersuchungen der Komplexbildung zwischen Cyclin T1 und Tat ergaben, dass ein ternärer Komplex zusammen mit Hexim1 nicht gebildet werden kann, sondern dass vielmehr Tat Hexim1 von seiner Bindungsstelle an Cyclin T1 verdrängt [3]. Diese wechselseitig exklusive Bindung hebt den P-TEFb vermittelten Transkriptionsarrest auf und führt zu einer HIV-stimulierten Transkriptionsaktivierung (Abb. 4).
Verdrängungs-Kompetitionsexperimente mittels schneller Fluoreszenzkinetik zeigten darüber hinaus eine über 10fach erhöhte Bindungsaffinität des Tat-Proteins an Cyclin T1 im Vergleich zu Hexim1. Dabei ist neben der reduzierten Dissoziationsrate für die Cyclin T1-Tat-Interaktion auch die Assoziation im Vergleich zu Hexim1 verlangsamt. Diese Beobachtung spricht für eine induzierte Faltung des intrinsisch unstrukturierten Tat-Proteins bei der Bindung an Cyclin T1. Zukünftige Arbeiten der Dortmunder Arbeitsgruppe konzentrieren sich auf die Einbeziehung der Kinase Cdk9 sowie verschiedener RNA-Fragmente von 7SK und TAR in die Experimente zur Analyse des inhibitorischen P-TEFb/Hexim1/7SK-Komplexes und des transkriptionsaktiven P-TEFb/Tat/TAR-Komplexes.




