Neuartige chemische Sonden in der Autophagozytose-Forschung
Forschungsbericht (importiert) 2014 - Max-Planck-Institut für molekulare Physiologie
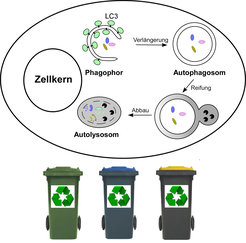
Autophagozytose
In unserer Gesellschaft muss der Abfall sortiert und wiederaufbereitet werden, damit er keinen Schaden anrichten kann. In Analogie dazu dient die Autophagozytose in der eukaryotischen Zelle als während der Evolution konservierter Selbstverdauungsprozess dazu, überschüssige oder beschädigte Organellen und langlebige bzw. verklebte Proteine zu entfernen. Wenn Zellen hungern oder gestresst werden, verdauen sie zytoplasmatische Komponenten mittels Autophagozytose, um sich mit Nährstoffen für das Überleben zu versorgen (Abb. 1).
Dieser Vorgang erfordert die Bildung von Vesikeln mit einer Doppelmembran, den Autophagosomen, die zytoplasmatische Komponenten abkapseln und sich einverleiben. Anschließend übergeben sie ihre Ladung anderen Organellen, den Lysosomen, die sie dann verdauen. So versteht man, dass die Autophagozytose eine kritische Rolle in der normalen Zellphysiologie spielt. Bei gestörter Funktion ist sie an verschiedenen menschlichen Krankheiten beteiligt, an Krebs und Degeneration der Nerven sowie an Infektionen und Alterungsprozessen [1; 2]. Agenzien, die die Autophagozytose induzieren, könnten sich bei der Behandlung der Parkinson- und der Huntington-Krankheit wirksam zeigen, indem sie mutierte Proteine, die zur Aggregatbildung neigen, eliminieren. Wird die Autophagozytose in Krebszellen, die sie unter Stress aktivieren, unterdrückt, könnte sich das in einer Krebstherapie als förderlich erweisen.
Chemische Proteinmodifikation
Die Autophagozytose-Forschung hat ein vitales Interesse am Studium der Proteinfunktionen, sowohl im Reagenzglas als auch in lebenden Zellen und Organismen. Sind Proteinstrukturen auf der Ebene der Transkription vom Genom und der Translation in den Ribosomen grundsätzlich einfach, so werden sie durch posttranslationale Veränderungen – wie die Addition von Phosphatresten, die Verknüpfung mit Fettsäuren, Zuckern und weiteren niedermolekularen Resten – derart komplex, dass ihre Synthese durch molekularbiologische Rekombination eine große Herausforderung darstellt, was die Homogenität und die Ausbeute betrifft. Um die Funktion von Proteinen in vitro und in vivo zu untersuchen, müssen sie also chemisch modifiziert werden. So lassen sich nicht nur posttranslationale Veränderungen erzeugen, sondern auch synthetische Sonden wie Fluorophore, Affinitätsmarker und weitere Marker in Proteine einführen [3].
Ligation von Proteinen
Die Entwicklung von zwei chemischen Methoden, Proteinreste miteinander zu verknüpfen, haben in den letzten Jahren zu Erfolgen bei der Synthese modifizierter Proteine geführt: die Native Chemische Ligation (NCL) und die Anwendung von selektiven chemischen Reaktionen zur Markierung, die die biologische Funktion der Proteine nicht stören (bioorthogonale Chemie) [4]. Forscher am Max-Planck-Institut in Dortmund haben einen ganzen Satz von effizienten Ligationsmethoden entwickelt, zu denen neben der NCL die Click-Ligation, die Ligation via Oxime und die Affinitäts-Konjugation zählen. So konnten nicht nur posttranslationale Modifikationen erzeugt werden, sondern es gelang auch mit Hilfe von einfach-, zweifach- oder dreifachmarkierten Proteinen, Protein-Protein-Wechselwirkungen sowie die Faltung und Entfaltung von Proteinen zu untersuchen. Selbst die Aktivierung von Enzymen in Echtzeit und der „Verkehr“ der Proteine in lebenden Zellen ließen sich mit diesen Techniken studieren [5-9].
Lipidierung von Proteinen
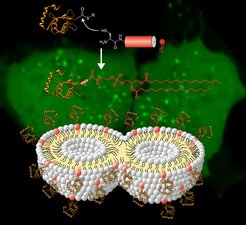
Abb. 2: Im oberen Bildteil wird die Synthese von lipidiertem LC3-Protein in einer Kombination aus Peptidsynthese mit Lipidrest und Expressed Protein Ligation gezeigt. Die Rolle, die lipidiertes LC3-Protein bei der Verschränkung und der Fusion der Membranen spielt, wird im unteren Bildteil illustriert.
Bei der Bildung von Autophagosomen spielt eine Proteinfamilie eine Schlüsselrolle, die als leichte Kette 3 (LC3) in Proteinen vorkommt, die sich mit Mikrotubuli in der Zelle assoziieren. Um korrekt und funktionell in der Membran verankert zu werden, wird das Protein in der Zelle am Carboxyterminus mit einem Lipid, dem Phosphatidylethanolamin, modifiziert. Wie LC3 auf molekularer Ebene die Biogenese der Autophagosomen steuert, ist zur Zeit weitgehend unklar. In Experimenten mit künstlich lipidiertem LC3 lässt sich eine Teilantwort finden. Dazu wurde der aminoterminale Teil von LC3 in Escherichia coli exprimiert und isoliert. Das carboxyterminale lipidierte Peptid wurde durch Synthese gewonnen. Nach Ligation beider Teile erhielten die Forscher ein biologisch aktives LC3 (LC3-PE) in einer Ausbeute von mehreren Milligramm (Abb. 2).
Die Forscher am MPI für molekulare Physiologie haben herausgefunden, dass LC3-PE die Verkettung und Fusion von Membranen bei physiologischen Konzentrationen von Phosphatidylethanolamin fördert, was für eine Beteiligung von LC3-PE bei der Elongation der Autophagosomen spricht [8].
Positive Störung durch kleine Moleküle
Biologische Systeme bestehen aus einem Netzwerk interagierender Biomoleküle. In der Antwort auf extra- oder intrazelluläre Stimuli sind sie raum-zeitlich reguliert. Die Chemische Genetik nutzt kleine Moleküle, um Proteinaktivitäten zu ändern, und stellt eine robusten Ansatz dar, die Dynamik biologischer Systeme aufzuklären. Sie ist schnell, reversibel und skalierbar. Deshalb ist die Chemische Genetik in der Lage, die Komplexität biologischer regulatorischer Netzwerke aufzulösen. Klassische genetische Verfahren wie Gen-Knockout oder RNA-Interferenz können das nicht leisten. Um potente Modulatoren (kleine Moleküle, die am Prozess beteiligte Makromoleküle beeinflussen) der Autophagozytose zu identifizieren, durchsuchten die Forscher eine große Substanzbibliothek. Nicht nur die Identifizierung von potentiellen Modulatoren, sondern auch die Identifizierung ihrer Zielmoleküle werden neue Einsichten in die Regulationsmechanismen der Autophagozytose erlauben. Unter diesen kleinen Molekülen sind auch solche, die als Startsubstanzen für die Entwicklung von Therapien dienen; sie modulieren die Autophagozytose und können damit eine Behandlung von Krebs oder neurodegenerativen Erkrankungen ermöglichen.
Schalter für Proteinstrukturen
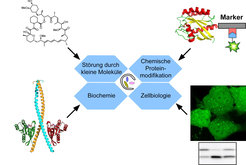
Die Forscher am Institut haben ein robustes System entwickelt, mit dem zelluläre Abläufe im positiven Sinn durch kleine Moleküle gestört werden können. So lässt sich z. B. die Dimerisierung, d. h. die nicht kovalente Verknüpfung von zwei Proteinkomponenten zu einem neuen funktionstragenden Makromolekül, chemisch durch kleine Moleküle induzieren. Der Prozess arbeitet wie ein Schalter, mit dem die Forscher die Funktion des Proteins in lebenden Zellen an- und abschalten können. Die sofort einsetzende Wirkung der Substanz, die die Dimerisierung hervorruft, kann sehr schnell durch die Zugabe der Substanz, die die Dimerisierung aufhebt, gestoppt werden. Und dies geschieht in Sekunden oder Minuten, wodurch sich die tatsächliche Dynamik gewisser biologischer Prozesse verfolgen lässt [10]. Die Beschränkungen traditioneller genetischer Methoden werden in solchen Ansätzen aufgehoben (Abb. 3).
Auch die Mechanismen, die bei der Bildung der Autophagosomen eine Rolle spielen, lassen sich in solchen Systemen studieren.
Literaturhinweise
proteins


