Nie die Kontrolle verlieren: Das Kinetochor am Kontrollpunkt der Zellteilung
Forschungsbericht (importiert) 2015 - Max-Planck-Institut für molekulare Physiologie
Einführung
Proteine sind die Bausteine des Lebens und durch ihren Zusammenbau, einem LEGO-Spiel vergleichbar, entstehen überraschend komplexe Strukturen in den Zellen unseres Körpers. Eine dieser Strukturen ist das Kinetochor, eine komplexe makromolekulare Maschine, die aus circa 30 Kernuntereinheiten, jede in vielfacher Kopie, und einer großen Zahl regulatorischer Untereinheiten besteht (Abb. 1).
Kinetochore sind das molekulare Klettband, das die Interaktion von Chromosomen mit der Kernspindel während der Zellteilung (Mitose) ermöglicht. Kinetochore binden an die Mikrotubuli der Kernspindel, dynamische Polymere des Proteins Tubulin. Die Fähigkeit des Kinetochor-Klettbands, Kontakt mit den Mikrotubuli herzustellen, ist die Voraussetzung für die Chromosomenverteilung auf die Tochterzellen und damit letztendlich für die Existenz des Lebens über Generationen hinweg.
Kinetochore haben aber eine weitere und damit verwandte fundamentale Funktion: sie kontrollieren die Genauigkeit der Chromosomentrennung, indem sie die Komponenten eines Signalwegs bereitstellen, der als spindle assembly checkpoint (SAC) bezeichnet wird. Der SAC ist eine Art Kontrollpunkt: Er besitzt einen Rückkopplungsmechanismus, der bewirkt, dass der nächste Mitoseschritt nur starten kann, wenn alle Chromosomen korrekt an der Kernspindel angeheftet sind. Die Forscher am MPI für molekulare Physiologie haben das Ziel, den Kinetochor-Spindel-Komplex und die SAC-Funktion in vitro aus gereinigten Komponenten zusammenzusetzen, um so die tieferen Zusammenhänge dieses lebenswichtigen Prozesses zu begreifen.
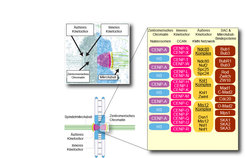
Abb. 1: Organisation des Centromers und des Kinetochors.
Kinetochore sind spezialisierte Strukturen am Centromer, die für den reibungslosen Prozess der Chromosomentrennung auf die Tochterzellen erforderlich sind.
Legende:
violett: Centromer-Chromatin
pink: Inneres Kinetochor (liegt direkt dem
Centromer-Chromatin an)
gelb: Äußeres Kinetochor (an der Mikrotubuli-
Bindung beteiligt)
braun: Äußere Lage der SAC-Proteine und
zusätzlicher Regulatoren der Mikrotubulus-
Bindung
Die einzelnen Proteine sind im Text bezeichnet.
Die Mitose muss mit kontrollierter Genauigkeit arbeiten, damit in unseren Zellen keine Abweichungen vom normalen Gehalt an genetischer Information auftreten. Eine Ungleichverteilung geht einher mit Tumorbildung und ist der Grund angeborener Defekte wie z. B. des Down-Syndroms.
Strukturelle Organisation des inneren und äußeren Kinetochors
Inneres Kinetochor
Für die Untersuchung biologischer Funktionen ist die biochemische Wiederherstellung eines Proteinkomplexes in vitro von großer Bedeutung. So erlaubt sie beispielsweise einen Einblick in die physikalische Interaktion von Proteinen und deren Regulation, z. B. durch posttranslationale Modifikationen wie die Verknüpfung mit Phosphatresten. Sie ist auch essenziell für die Anwendung bestimmter Analysetechniken wie Röntgenkristallographie oder Kryo-Elektronenmikroskopie, die Strukturinformationen von Proteinen und Proteinkomplexen liefern.
Mit diesen Techniken gelang es, strukturelle und funktionelle Informationen über mehrere Subkomplexe des sog. „konstitutiven zentromerassoziierten Netzwerks“ (CCAN in Abb. 1) zu gewinnen. Es konnte gezeigt werden, dass CENP-M, eine kleine Untereinheit von CCAN, notwendig ist, einen Komplex aus vier Untereinheiten (den CENP-HIKM Komplex) zu stabilisieren [1]. Auch die direkte physikalische Interaktion zwischen CENP-C, einer anderen CCAN- Untereinheit, und dem CENP-HIKM-Komplex konnte nachgewiesen werden [2]. Zusammen mit früheren Arbeiten der Forscher [3] bot diese Entdeckung die Möglichkeit, für die Funktion von CENP-C das Konzept einer „Blaupause“ (als Instruktionsplan) für den Zusammenbau des Kinetochors aufzustellen.
Äußeres Kinetochor
Auch beim Studium des äußeren Kinetochors wurden Fortschritte gemacht. Es besteht aus drei verschiedenen Subkomplexen mit zehn Untereinheiten, den Knl 1-, Mis12- und Ndc80-Komplexen (KMN-Netzwerk, gelb in Abb. 1), die zusammen die Schnittstelle für die Mikrotubulus-Bindung darstellen. An dieser Schnittstelle wird auch der SAC kontrolliert, und es ist deshalb von immenser Bedeutung, seine Regulation zu verstehen. Der Mis12-Komplex ist der Dreh- und Angelpunkt im KMN-Netzwerk: Er ermöglicht dem ganzen Netzwerk, mit den darunter liegenden Teilen des inneren Kinetochors zu interagieren und bringt die anderen Komponenten des KMN-Netzwerks geordnet zueinander [4]; [3]. Die Schnittstelle für die Mikrotubulus-Bindung liegt auf den Ndc80-Untereinheiten, die kürzlich biochemisch und strukturell in ausgezeichneten Details charakterisiert worden sind [5]; [6].

Abb. 2: Strukturanalyse der C-terminalen Region von Knl1.
Knl1 ist eine Untereinheit, die eine entscheidende Rolle in der Regulation des "spindle assembly checkpoints" (SAC) spielt. SAC ist ein wichtiger Kontrollpunkt in der Mitose.
A) Tandem- RWD-Domänen (RWD-N und RWD-C)
B) Ihre Interaktion mit einem Segment aus dem C-terminalen Teil von Nsl1 (Mis12-Komplex)
C) Eine Punktmutation in Knl1 an der Schnittstelle mit Nsl1 verhindert die Verbindung mit dem Kinetochor in einem Knl1-GFP-Reporter-Konstrukt
Bezeichnungen im Bild:
GFP: Grünes Fluoreszierendes Protein
GFP-WT: GFP-Knl1-Konstrukt Wildtyp
GFP-S2169E: GFP-Knl1-Konstrukt mit Austausch von Serin 2169 gegen Glutaminsäure
CREST: CREST Syndrom Antisera gegen CENPs
DAPI: DNS-färbender Fluoreszenzfarbstoff
Die beiden Untereinheiten Knl1 und Zwint im Knl1-Komplex spielen eine entscheidende Rolle in der Regulation des spindle assemby checkpoints. Das humane Knl1 ist ein sehr großes Protein, für das die Strukturrechnung zeigt, dass großen Teilen eine definierte Tertiärstruktur fehlt und das Protein somit als intrinsisch ungeordnet zu bezeichnen ist. Einzig für die C-terminale Region wird eine globuläre Struktur vorhergesagt, aber die genaue Struktur war bisher unbekannt. In massenspektrometrischen Analysen von quervernetzten Proteinen wurde eine Bindestelle der C-terminalen Domäne von Knl1 auf dem C-terminalen Ende der Mis12-Untereinheit Nsl1 identifiziert [4]. In der Kristallstruktur der C-terminalen Domäne von Knl1 zeigte sich eine strukturelle Verwandtschaft mit anderen Proteinen des Kinetochors, vor allem mit den auf den CCAN gerichteten Domänen des Ndc80-Komplexes [7]. Die C-terminale Region von Knl1 besteht aus zwei hintereinander angeordneten Strukturen, den RWD-Domänen, die an Protein-Protein-Interaktionen beteiligt sein sollen (Abb. 2A). Um dies zu untersuchen, wurde ein synthetisches Peptid, das die Sequenz des Bereichs von Nsl1 besitzt, der für die Anheftung von Knl1 am Kinetochor verantwortlich ist, mit dem C-terminalen Ende von Knl1 kokristallisiert und röntgenkristallografisch untersucht. Tatsächlich fanden die Forscher, dass das Nsl1-Peptid einen „Grat“ in der Kopplungsstelle der beiden RWD-Domänen besetzt (Abb. 2B). In diesem Grenzbereich mutierte Proteine verhinderten die Anlagerung von Knl1 an das Kinetochor, was die Bedeutung dieser Interaktion unterstreicht (Abb. 2C).
In Zusammenarbeit mit der Abteilung für Strukturelle Biochemie am MPI in Dortmund gelang es, eine erste dreidimensionale elektronenmikroskopische Struktur des KMN-Netzwerks zu erhalten (Abb. 3) [7]. Bereits vorab identifizierte Positionsmarker auf dem KMN-Netzwerk, wie mögliche Bindungsstellen für SAC, konnten Bereichen auf der dreidimensionalen Struktur zugeordnet werden.

Abb. 3: Domänenzuordnung in der 3D-Rekonstruktion des KMN-Netzwerks und relative Position der Signalkomplexe des Kontrollpunkts
A) Molekulares Modell des Ndc80-Komplexes im KMN-Netzwerk
B) Molekulares Modell der Knl1-C-terminalen RWD-Domäne im KMN-Netzwerk
C) Schema des äußeren Kinetochors mit relativen Positionen der MELT-Wiederholungen und der stufenweisen Anordnung der Proteinuntereinheiten
Funktionelle Organisation der mit dem Kontrollpunkt interagierenden Elemente von Knl1
Für die Anbindung an SAC-Proteine gibt es zwei Arten von Aminosäuremotiven in Knl1: erstens das KI-Motiv mit den Aminosäuren Lysin (K) und Isoleucin (I), das auch in anderen Wirbeltieren gefunden wurde, und zweitens das MELT-Motiv aus den Aminosäuren Methionin (M), Glutaminsäure (E), Leucin (L) und Threonin (T). KI interagiert mit einem Motiv aus 34 Aminosäureresten (Tetratricopeptid, TRP) auf den Kontrollpunkt-Proteinen Bub1 und BubR1. MELT, das in der Evolution ubiquitär vorkommt, rekrutiert einen SAC-Komplex aus Bub1- und Bub3-Proteinen, in einem Prozess, der von der Verknüpfung mit Phosphatresten abhängig ist. All diese Motive befinden sich in der ersten Hälfte von Knl1. Da die genaue Beteiligung von KI- und MELT-Motiven an der Signaltransduktion durch SAC und der Ausrichtung der Chromosomen unklar war, konnte gezeigt werden, dass KI-Motive eng mit dem benachbarten einfachen MELT-Motiv in den N-terminalen 250 Resten des humanen Knl1 kooperieren, um den kompletten Zusammenbau der SAC-Proteine zu steuern [8]. In Zellen, die von endogenem Knl1 befreit worden waren, reichte der auf den Kinetochor gerichtete N-terminale, 250 Reste umfassende Teil von Knl1 aus, um die Ausrichtung von SAC und den Chromosomen wiederherzustellen. Individuelle Wiederholungen von MELT außerhalb dieser Region, denen flankierende KI-Motive fehlen, gehen qualitativ ähnliche Interaktionen ein, aber mit geringerer Effizienz. MELT-Sequenzen auf Knl1 scheinen also die Plattform zu sein, auf der SAC-Komplexe zusammengebaut werden, wohingegen KI-Motive als Verstärker der MELT-Funktion bei diesem Zusammenbau auftreten können.
Die Rolle des SAC-Proteins Bub3 als Modul für die Rekrutierung des Kinetochors
Bevor die Anbindung der Bub1:Bub3-Komponenten des SAC an das Kinetochor durch die MELT-Sequenzen erfolgen kann, müssen diese vorab von der Kinase Mps1, einer unabdingbaren Komponente des SAC, mit Phosphatresten modifiziert werden. Um zu verstehen, wie diese veränderten MELT-Motive erkannt werden, wurde der Komplex in vitro mit gereinigten Proteinen rekonstituiert [9]. Es zeigte sich, dass Bub3, ein Propeller mit sieben Blättern aus beta-Faltblattstrukturen, die MELT-Motive „liest“ (Abb. 4).
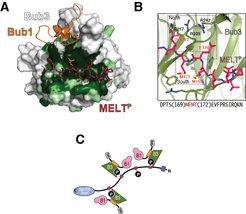
Abb. 4: Anheftung des Bub1/Bub3-Komplexes am Kinetochor
A) Bub3 ist ein siebenblättriger ß-Faltblatt-Propeller. Bereiche in weiß mit niedriger, in hellgrün mit höherer und dunkelgrün mit hoher evolutionärer Sequenzhomologie. Eingefügt sind bindungsspezifische Abschnitte von Bub1 (orange) und mit Phosphatresten modifiziertem MELT (MELTp) (rot).
B) MELTp liegt in der Schnittstelle mit der höchsten Sequenzhomologie. Die MELTp-Bindestelle in Bub3 enthält mehrere positiv geladene Aminosäurereste (Arginine 217, 239 und 242).
C) Schematische Übersicht für die Rekrutierung des Bub1/Bub3-Komplexes durch MELTp während der Signalgebung am Kontrollpunkt
Bezeichnungen in Abb. 4 C)
Rote Rechtecke: MELT
P: Phosphatreste
B1: Bub1
B3: Bub3
TPR: TetratricopeptidIn der Kristallstruktur des Bub3-Bub1-MELT-Komplexes enthält Bub3 eine außergewöhnlich gut konservierte Bindestelle, die an die modifizierten MELT-Sequenzen auf der Seite des beta-Faltblatt-Propellers in einer bisher unbekannten Weise andockt. Mutationen in der Bindestelle von Bub3 verhindern, dass das Kinetochor die SAC-Kinase Bub1 rekrutieren kann. Äußerst wichtig ist auch, dass solche Mutationen einen Defekt am Kontrollpunkt auslösen. Die spezifische Erkennung der mit Phosphatresten modifizierten Zielstrukturen durch Bub3 garantiert also die korrekte Signalweiterleitung am Kontrollpunkt.
In einer Nachfolgestudie [10] wurde charakterisiert, wie Bub1 die Erkennung von modifizierten MELT-Motiven moduliert. Eine kurze Region von Bub1, die eine „Schleife“ bildet, ist notwendig, um die Bindungsaffinität des Bub1:Bub3-Komplexes zu den MELT-Sequenzen zu verstärken. Diese überraschend kurze Sequenz sorgt für den Unterschied zwischen Bub1 und BubR1, die als Produkte einer Genverdopplung sehr eng verwandte Aminosäuresequenzen haben. BubR1 besitzt eine andere Schleifensequenz, die nicht an der Verstärkung der Bindung von Bub3 an MELT-Sequenzen beteiligt ist.
Ausblick
Die hier vorgestellten Ergebnisse ließen sich nur durch Arbeiten auf verschiedenen Ebenen erreichen, die von der physikalischen Organisation des Kinetochors bis zur delikaten Regulation bei der Rekrutierung der SAC-Proteine gehen. Dies spiegelt sich ebenfalls in der Anwendung verschiedenster Techniken aus der Biochemie (Rekonstitution), der Strukturbiologie (Röntgenkristallographie und Elektronenmikroskopie) sowie der Zellbiologie (funktionelle Validierung) wider. Diese Vorgehensweise scheint zunächst einmal nur kleine Fortschritte zu bringen, aber sie geduldig zu verfolgen, wird es möglich machen, die essenziellen Einzelheiten dieses fundamentalen biologischen Prozesses aufzuklären.



