Wie sich Peroxisomen ihre Enzyme angeln
Experten verschiedener Fachgebieten enthüllen gemeinsam die erste Struktur des peroxisomalen Andock-Apparates
Schaut man in das Innere einer Zelle, finden sich zahlreiche Bläschen unterschiedlicher Größe. Diese sogenannten Organellen sind von einer Membran abgeschlossene Reaktionsräume, in denen bestimmte Aufgaben durch Enzyme erfüllt werden: Während in den Mitochondrien, den Kraftwerken der Zelle, Energie gewonnen wird, arbeiten in den Peroxisomen, die auch als Entgiftungsapparate der Zelle beschrieben werden, dutzende Enzyme z.B. am Abbau von schädlichen Fettsäuren oder der Synthese essentieller Lipide. Dabei fällt das giftige Wasserstoffperoxid an, das in den Peroxisomen neutralisiert wird. Defekte der Peroxisomen oder ihrer Enzyme führen zu schweren Erkrankungen mit meist tödlichen Folgen.
Signalsequenzen leiten Enzyme zum richtigen Ziel
Die meisten Organellen, wie auch die Peroxisomen, können ihre Enzyme nicht selbst bilden. Diese werden an den Ribosomen der Zelle produziert und müssen anschließend korrekt auf die jeweiligen Organellen verteilt werden. Dafür tragen die peroxisomalen Enzyme eine von zwei spezifischen Signalsequenzen, die von Importrezeptoren im Cytosol erkannt werden. Die Gruppe um Ralf Erdmann, konnte bereits in früheren Arbeiten zeigen, dass für jede der beiden Signalsequenzen eine extra Pore existiert, die den beladenen Rezeptor in den Innenraum der Peroxisomen schleust. „Bisher sind jedoch nur wenige Details über diesen Prozess bekannt,“ sagt Erdmann, der schon seit über 30 Jahren an Peroxisomen forscht.
Eine zentrale Rolle wird hierbei einem Andock-Komplex zugeordnet, der die beladenen Rezeptoren zu der peroxisomalen Membran dirigiert und sie dort einfängt. Wie dieser Komplex mit Hilfe der Proteine Pex13p, Pex14p und Pex17p gebildet wird und im Detail funktioniert, ist aber noch rätselhaft. Nun ist es dem Team um Christos Gatsogiannis mittels Elektronenmikroskopie zum ersten Mal gelungen, die Architektur des Komplexes zu bestimmen und so neue mechanistische Erkenntnisse zu erlangen.
„Die enge Zusammenarbeit von Wissenschaftlerinnen und Wissenschaftlern mit Expertisen aus verschiedenen Fachgebieten war Schlüssel zum Erfolg“, betont Bettina Warscheid.
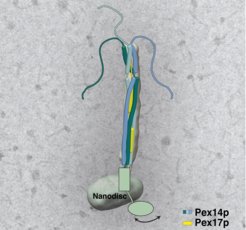
Nach Enzymen angeln
Tobias Hansen ist es im Rahmen seiner Doktorarbeit an der Universität Bochum gelungen einen hochreinen Komplex der Proteine Pex14p und Pex17p zu isolieren. Mit nativer Massenspektrometrie konnte Daniel Wendscheck, Doktorand an der Universität Freiburg, feststellen, dass sich der Komplex insgesamt aus drei Kopien des Proteins Pex14p und einem Pex17 zusammensetzt. Pascal Lill, Doktorand am MPI in Dortmund, rekonstituierte den Andock-Komplex in einer nahezu nativen Membranumgebung um die Bedingungen an der peroxisomalen Membran zu simulieren und gewann mit Hilfe der Kryo-Elektronenmikroskopie und -tomographie die Struktur des Komplexes (Bild 1). Sie zeigt längliche, parallel zueinander angeordnete Stränge, die zusammen eine stabähnliche Gesamtstruktur ausbilden. Dieser „Stab“ schaut mit etwa zwei Dritteln aus den Peroxisomen heraus. Durch weitere Experimente konnten die drei Gruppen in einem spannenden Prozess schließlich auch die genaue Anordnung der Proteine aufklären. Am Ende der Struktur zeigen die drei Pex14p Proteine flexible Bereiche, die wie Tentakel in den Raum der Zelle herausragen. Die Wissenschaftler vermuten, dass die Fangarme dazu dienen, möglichst viele beladene Rezeptoren zu angeln (Bild 2).

Christos Gatsogiannis erklärt: „Dieser Komplex ist von essentieller Bedeutung für funktionstüchtige Peroxisomen. Unser erstes Bild seiner Struktur und ein mechanistisches Modell seiner Funktion bilden eine wichtige Grundlage für weitere Untersuchungen der Peroxisomen und könnten auch zu einem besseren Verständnis peroxisomaler Krankheiten beitragen.“
Die ProtagonistInnen des Projekts
Bettina Warscheid ist Leiterin der Abteilung Biochemie und Funktionelle Proteomforschung am Institut für Biologie II der Universität Freiburg. Ralf Erdmann ist Leiter der Abteilung Systembiochemie am Institut für Biochemie und Pathobiochemie der Universität Bochum. Christos Gatsogiannis (bis vor Kurzem am MPI für molekulare Physiologie in Dortmund) hat im September einen Ruf als Professor für Elektronen-Tomographie nanostrukturierter Systeme am Institut für medizinische Physik und Biophysik der medizinische Fakultät Münster angenommen und wird bald seine Gruppe am neuen „Center for Nanoscience“ der Westfälischen Wilhelms-Universität Münster etablieren. Die Zusammenarbeit erfolgte im Rahmen der von der Deutschen Forschungsgemeinschaft geförderten Forschergruppe „Struktur und Funktion des peroxisomalen Translokons“.

