Wie Zellen ihre Umgebung durch Zellkontraktion ertasten
Optogenetische Untersuchungen der Dehmelt Gruppe entschlüsseln die Regulation der Zellkontraktion
Körperzellen können ihre Umgebung auf zweierlei Weise wahrnehmen: chemisch und physikalisch. Letzteres wird gewährleistet durch ein aktives „Ertasten“ der Umgebung. Die dazu notwendige Veränderung der Zellform wird auf molekularer Ebene durch pulsierende Kontraktionen hervorgerufen. Ein komplexes Netzwerk aus Signalmoleküle reguliert diesen „mechanischen Tastprozess“, der eine zentrale Rolle z. B. in der Embryonalentwicklung oder dem Tumorwachstum spielt. So entwickeln sich Stammzellen - die Alleskönner unter den Zellen – in einer „tastbar“ harten Umgebung eher zu Knorpel- oder Knochenzellen und in einer „tastbar“ weichen Umgebung beispielsweise eher zu Muskel- oder Nervenzellen.
Für die biomedizinische Forschung ist es von großem Interesse, herauszufinden, wie diese pulsierenden Kontraktionen in Zellen zeitlich und räumlich durch die Signalweiterleitung in Netzwerken reguliert werden.
Signalmoleküle gezielt mit Licht stören
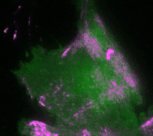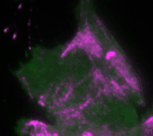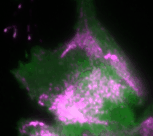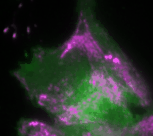
In der aktuellen Studie des Teams um Leif Dehmelt wurde das Signalnetzwerk, das die Zellkontraktion steuert, durch gezielte Störung einzelner Signalproteine manipuliert. Dazu werden die Proteine genetisch so verändert, dass sie durch das Bestrahlen mit Licht einer bestimmten Wellenlänge aktiviert oder deaktiviert werden können. Gleichzeitig wird die Reaktion des Signalnetzwerks und damit die Antwort des biologischen Systems gemessen. So können Rückschlüsse auf die Signalverarbeitung durch dieses Netzwerk gezogen werden. Basierend auf diesen Messergebnissen wurde ein theoretisches Modell erstellt, das die Komponenten (Signalmoleküle) und ihre Interaktion im Signalnetzwerk räumlich und zeitlich beschreibt. Eine zentrale Rolle im Signalnetzwerk spielen sogenannte Rückkopplungsschleifen. Diese entstehen, wenn ein weitergeleitetes Signal im Netzwerk den Ausgangspunkt der Signalübertragung beeinflusst. Dies kann das Signal entweder verstärken oder abschwächen. Die Wissenschaftler konnten zeigen, dass ein Zusammenspiel dieser Rückkopplungsschleifen hochdynamische Zellkontraktions-Pulse erzeugt. Das theoretische Modell erlaubt auch Vorhersagen, wie das Signalnetzwerk reagiert, wenn Rückkopplungsschleifen gestört werden. Die theoretischen Ergebnisse konnten experimentell in einzelnen lebenden Zellen durch licht-basierte, fein abgestimmte Störungen des Signalnetzwerkes bestätigt werden.
Echtzeit-Kontrolle von Signalnetzwerken
Mithilfe dieser als „Optogenetic Tuning“ bezeichneten Technik, konnten die Wissenschaftler zum ersten Mal zeigen, dass das Signalnetzwerk besonders intensive, und regelmäßige Pulse des Zellkontraktions-Regulators Rho bei mittleren Konzentrationen des Rho Aktivators GEF-H1 erzeugt (Video 1). Hierdurch produzieren Zellen ausgeprägte Kontraktions-Pulse, welche eine wichtige Rolle in der Embryonalentwicklung spielen. Das Signalnetzwerk ist hingegen bei niedrigen Konzentrationen des Aktivators GEF-H1 besonders empfindlich, um schwache mechanische oder biochemische Signale zu verstärken.
„Durch unsere Studien haben wir ein grundlegendes Verständnis entwickelt, um die Regulation der Zellkontraktion auch in komplexeren biologischen Systemen wie Geweben zu untersuchen. Dies könnte dazu beitragen, die Rolle der Zellkontraktion in krankheitsrelevanten Prozessen aufzuklären“, erklärt Leif Dehmelt.
