Stefan Raunser
Direktor, Strukturbiochemie
Aktuelles und Pressemitteilungen
Forschung im Überblick
> Zur Abteilung Strukturbiochemie
Unsere Forschung konzentriert sich auf das molekulare Verständnis fundamentaler zellulärer Prozesse. Zum einen interessieren wir uns für die Membranhomöostase in eukaryotischen Zellen, zum anderen wollen wir die molekularen Grundlagen der Muskelkontraktion verstehen. Ein weiterer Schwerpunkt liegt auf dem molekularen Verständnis des Wirkmechanismus bakterieller Toxine.
Unser übergeordnetes Ziel ist es, die Mechanismen, die diesen Prozessen im gesunden und kranken Organismus zugrunde liegen, im molekularen Detail zu verstehen. Dafür ist es unabdingbar, die Struktur, und damit die Funktion der beteiligten Proteine und Proteinkomplexe aufzuklären.
Zur Struktur- und Funktionsanalyse nutzen wir Elektronenkryomikroskopie (cryo-EM) und Elektronenkryotomography (cryo-ET), fluoreszenzbasierte Funktionsanalysen und ortsspezifische Mutagenese. Zur biophysikalischen Charakterisierung verwenden wir ITC, ESI-MS, Biolayer-Interferometrie, Thermophorese und CD-Spektroskopie.
Das Potenzial der Kryo-Elektronentomographie
Wir verwenden Kryo-Elektronenmikroskopie (Kryo-EM) und Kryo-Elektronentomographie (Kryo-ET), um die Grundlagen der Muskelkontraktion zu erforschen und Muskelerkrankungen besser zu verstehen. Insbesondere verwenden wir die Kryo-ET, um zu verstehen, wie Muskelproteine in ihrer zellulären Umgebung, d. h. in der Myofibrille, funktionieren. Sehen Sie sich dieses Video an, um zu erfahren, warum wir der Meinung sind, dass die Kryo-ET die strukturbiologische Schlüsseltechnik der Zukunft ist.
Scharfer Blick in die Muskelzelle
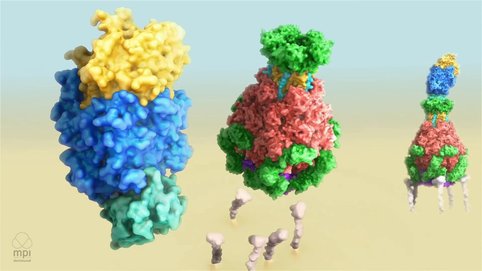
Mechanismus der Tc Toxin Giftinjektion
Ausgewählte Publikationen
Tamborrini D, Wang Z, Wagner T, Tacke S, Stabrin S, Grange M, Kho AL, Rees M, Bennet P, Gautel M, Raunser S (2023). Structure of the native myosin filament in the relaxed cardiac sarcomere. Nature
Quelle
Rice G, Wagner T, Stabrin M, Sitsel O, Prumbaum D, Raunser S (2023). TomoTwin: generalized 3D localization of macromolecules in cryo-electron tomograms with structural data mining. Nature Methods
Quelle
Oosterheert W, Klink B. U, Belyy A, Pospich S, Raunser S (2022). Structural Basis of actin filament assembly and aging. Nature
Quelle
Wang Z, Grange M, Pospich S, Wagner T, Kho A.L, Gautel M, Raunser S (2022). Structures from intact myofibrils reveal mechanism of thin filament regulation through nebulin. Science
Quelle
Wang Z, Grange M, Wagner T, Khoo AL, Gautel M, Raunser S (2021). The molecular basis for sarcomere organization in vertebrate skeletal muscle Cell
Quelle
Gatsogiannis C, Balogh D, Merino F, Sieber SA, Raunser S (2019). Cryo-EM structure of the ClpXP protein degradation machinery. Nat Struct Mol Bio
Quelle
Raisch T, Chang CT, Levdansky Y, Muthukumar S, Raunser S, Valkov E (2019). Reconstitution of recombinant human CCR4-NOT reveals molecular insights into regulated deadenylation. Nature Communications
Quelle
Gatsogiannis C, Merino F, Roderer D, Balchin D, Schubert E, Kuhlee A, Hayer-Hartl M, Raunser S. (2018). Tc toxin activation requires unfolding and refolding of a β-propeller. Nature
Quelle
Vinayagam D, Mager T, Apelbaum A, Bothe A, Merino F, Hofnagel O, Gatsogiannis C, Raunser S (2018). Electron cryo-microscopy structure of the canonical TRPC4 ion channel. eLife
Quelle
Klink BU, Zent E, Juneja P, Kuhlee A, Raunser S, Wittinghofer A. (2017). A recombinant BBSome core complex and how it interacts with ciliary cargo. eLife
Quelle
Pospich S, Kumpula EP, von der Ecken J, Vahokoski J, Kursula I, Raunser S. (2017). Near-atomic structure of jasplakinolide-stabilized malaria parasite F-actin reveals the structural basis of filament instability. Proc Natl Acad Sci U S A
Quelle
von der Ecken J, Heissler SM, Pathan-Chhatbar S, Manstein DJ & Raunser S (2016).Cryo-EM structure of a human cytoplasmic actomyosin complex at near-atomic resolution. Nature 534(7609):724-28.
Quelle
freely available!
Gatsogiannis C, Merino F, Serdiuk T, Prumbaum D, Roderer D, Leidreiter F, Meusch D, Müller DJ, and Raunser S (2016). Membrane insertion of a Tc toxin in atomic detail. Nature Structural and Molecular Biology 23(10):884-890.
Quelle
freely available!
Gatsogiannis C, Hofnagel O, Markl J, Raunser S (2015). Structure of Mega-Hemocyanin reveals protein origami in snails. Structure 23(1):93-103.
Quelle
Efremov R, Hofnagel O, Raunser S (2015). Architecture and Conformational Switch Mechanism of the Ryanodine Receptor. Nature 517(7532):39-43.
Quelle
freely available!
Whitney JC, Quentin D, Sawai S, LeRoux M, Harding BN, Ledvina HE, Tran BQ, Robinson H, Goo YA, Goodlett DR, Raunser S, Mougous JD (2015). An Interbacterial NAD(P)(+) Glycohydrolase Toxin Requires Elongation Factor Tu for Delivery to Target Cells. Cell 163(3):607-19.
Quelle
von der Ecken J, Müller M, Lehman W, Manstein DJ, Penczek PA, Raunser S (2015). Structure of the F-actin-tropomyosin complex. Nature 519(7541):114-7.
Quelle
freely available!
Meusch D, Gatsogiannis C, Efremov R, Lang A, Hofnagel O, Vetter I, Aktories K, Raunser S (2014). Mechanism of Tc toxin action revealed in molecular detail. Nature 508(7494):61-5.
Quelle
freely available!
Gatsogiannis C, Lang A, Meusch D, Pfaumann V, Hofnagel O, Benz R, Aktories K, Raunser S (2013). A syringe-like injection mechanism in Photorhabdus luminescens toxins. Nature. 495(7442):520-23
Quelle
freely available!